[참고] 본 자료에 사용된 제품은 SMART-Seq® v4 Ultra® Low Input RNA Kit for Sequencing이며, 이 제품은 SMART-Seq® mRNA로 변경되었습니다.
21세기 초 차세대 염기서열 분석(next-generation sequencing, NGS) 기술의 등장은 유전 연구에 접근하는 방식에 혁명을 일으켰다. 이후 대량 서열 분석 (bulk sequencing) 실험은 희귀 세포 유형에서 나타나는 중요한 생물학적 현상을 명확히 확인할 수 없다는 보고가 증가되면서, 연구자들은 개별 세포가 전체 시스템의 복잡한 생물학에 어떻게 기여하는지 더 잘 이해하기 위해 단일 세포 (single cell) 시퀀싱으로 전환하게 되었다.
Droplet 기반의 분석 방법(Droplet-based method)은 분석할 수 있는 세포 수가 많기 때문에, 부분적인 단일 세포 시퀀싱(single cell sequencing)에 많이 사용되어 왔다. 그러나 연구자들은 droplet 기반 방법만으로는 단일 세포가 가지고 있는 다양한 데이터들을 완전히 이해하기에 충분하지 않다는 것을 깨닫기 시작하였다. 우리가 최근
인터뷰한 캘리포니아 공과대학(California Institute of Technology)과 앨런 뇌과학연구소(Allen Institute of Brain Science)의 연구원들은
최근 연구 결과를 통해 SMART-Seq full-length sequencing과 droplet 기반의 분석 방법, 그리고 spatial scRNA-seq을 결합한 다각적 접근 방식이 특히 뇌에서 보다 효과적인 단일 세포 시퀀싱 (single cell sequencing)을 위한 보완적이고 균형 잡힌 접근 방식이라고 언급하였다. (Booeshaghi et al. 2020).
연구자들은 BRAIN Initiative Cell Census Network에서 만든 마우스 일차 운동 피질(mouse primary motor cortex, MOp) 샘플의 기존 시퀀싱 데이터를 분석하였다. 이 데이터는 6,000 여개 세포에 대한 full-length SMART-Seq v4(SMART-Seq) 데이터와 90,000여개 세포에 대한 10x Genomics Chromium(10xv3)에 대한 결과를 제공하였다(그림 1, Panel A). 두 기술 모두 광범위한 종류의 세포 유형을 식별할 수 있었으며, 보다 정교하게 분석한 결과, 10xv3는 분석한 세포 수가 더 많기 때문에 몇 가지 희귀 세포 유형을 식별하는 데 탁월한 반면, SMART-Seq의 더 높은 감도와 전장전사체 (full-length transcripts)의 프로빙은 알고 있는 세포 유형 분류 뿐 아니라 isoform의 정량도 가능하였다.
다음으로, 연구자들은 SMART-Seq v4로 분석한 glutamatergic neuron과 GABAergic neuron의 isoform 양(abundance)에 대한 differential analysis에 초점을 맞췄다. 두 세포 유형 모두에서 안정적인 발현을 보인 260개의 유전자에서 312개의 isoform 마커가 관찰되었으며,
H3f3b 유전자는 glutamatergic neuron에서 매우 높게 발현된 isoform중에 하나인 반면, GABAergic neuron에서는
H3f3b-204 isoform의 발현이 낮게 나타난 것을 확인하였다(원문 결과 참조). 저자는 "유전자 수준의 분석에서는 이와 같이 다양한 isoform 변화를 볼 수 없다"라고 언급하였다. SMART-Seq을 이용한 isoform의 정량, 세포 서브클래스(cell subclass) 분석 및 클러스터 할당(cluster assignments)은 모두 10xv3 데이터와의 비교를 통해 검증하였으며, 이러한 데이터들을 종합하여 MOp의 isoform atlas를 구출할 수 있었다(그림 2).
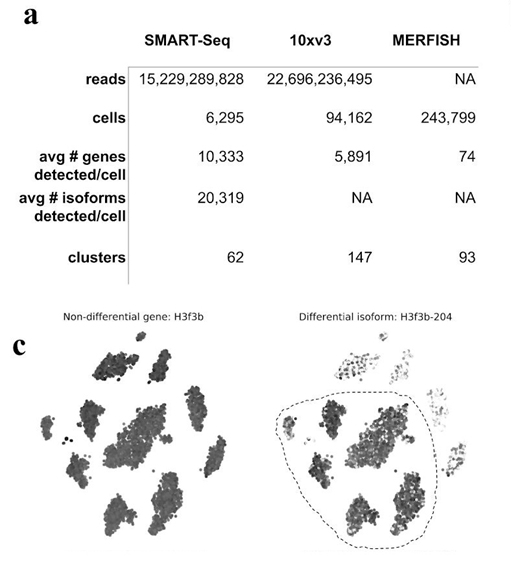
그림 1. 유전자 특이성을 보이지 않는 isoform specificity
Panel A. 분석한 결과 개요. Panel C. 세포 전체에 걸친
H3f3b 유전자 분포(좌) 및
H3f3b-204 isoform 분포(우).
※ 결과 출처 :
Booeshaghi et al. 2020 (일부 panel 제외),
Creative Commons Attribution 4.0 International License 하에서 사용.
 그림 2. Isoform atlas
Subclass에 따른 differential isoform marker를 나타낸 isoform atlas의 예시
그림 2. Isoform atlas
Subclass에 따른 differential isoform marker를 나타낸 isoform atlas의 예시
각 행은 하나의 subclass에 해당하고 각 열은 하나의 isoform을 나타낸다. SMART-Seq isoform 양(abundance)의 추정치는 TPM으로 표기하였으며, 최대 TPM은 해당 행의 클러스터에 특정 isoform 평균의 4배가 되도록 각 열의 크기를 조정하였다.
※ 결과 출처 :
Booeshaghi et al. 2020 (일부 panel 제외, 링크연결),
Creative Commons Attribution 4.0 International License 하에서 사용(링크연결).
Isoform 발현에 대한 이해를 더욱 높이기 위해, 다음으로 spatial MERFISH data에서 나온 데이터를 정밀하게 분석하기 위하여, SMART-Seq을 이용한 isoform정량 data를 이용하였다. 저자는
Pvalb 유전자를 면밀히 살펴보았고, MERFISH 결과를 통해 운동 피질 슬라이스(motor cortex slices)의 모든 층에 이 유전자가 퍼져 있음을 발견하였다. 그러나 SMART-Seq 데이터를 이용한 정량결과에서는 유전자의 두 가지 isoform 중 하나만 발현됨을 확인하였다. MERFISH에서 제공하는 유전자 수준 분석만으로 검사를 수행했다면, isoform의 이러한 공간적 발현 패턴(spatial expression)은 확인할 수 없었을 것이라고 언급하였다.
 그림 3. MOp 슬라이스의 모든 subclass에 있는 세포의 공간 위치. Pvalb cell은 별표로 표시
그림 3. MOp 슬라이스의 모든 subclass에 있는 세포의 공간 위치. Pvalb cell은 별표로 표시.
※ 결과 출처 :
Booeshaghi et al. 2020 (일부 내용 제외),
Creative Commons Attribution 4.0 International License 하에서 사용.
scRNA-seq analysis의 새로운 시대
아리스토텔레스는 "전체는 부분의 합보다 크다"고 말하였다. 이 연구는 연구자들의 질문에 접근하기 위해, 한 가지에만 집중하는 것보다 다양한 기술을 사용하여 이해를 훨씬 더 높은 수준으로 올릴 수 있는 방법을 보여주는 훌륭한 예이다. 특히 scRNA-seq의 경우 저자는 다음과 같은 관점을 제시하였다.
[SMART-Seq] 기술은 droplet 또는 spatial single-cell RNA-seq에 대한 경쟁자가 아닌 결과의 수준을 올릴 수 있는 보완물로 보아야 한다. 우리는 세포 유형을 식별하기 위해 droplet 기반 single-cell RNA-seq, isoform 분석을 위해 SMART-Seq[v4 chemistry], 마지막으로 SMART-Seq[v4 chemistry]에 의해 식별된 isoform 특이적 마커를 기반으로 하는 패널분석을 적용하는 spatial RNA-seq 으로 구성된 워크플로우를 제안한다. 이를 통해 다양한 기술의 강점을 효과적으로 활용할 수 있을 것이다."
참고문헌
Booeshaghi, A. S. et al. Isoform cell type specificity in the mouse primary motor cortex. bioRxiv 2020.03.05.977991 (2020).






